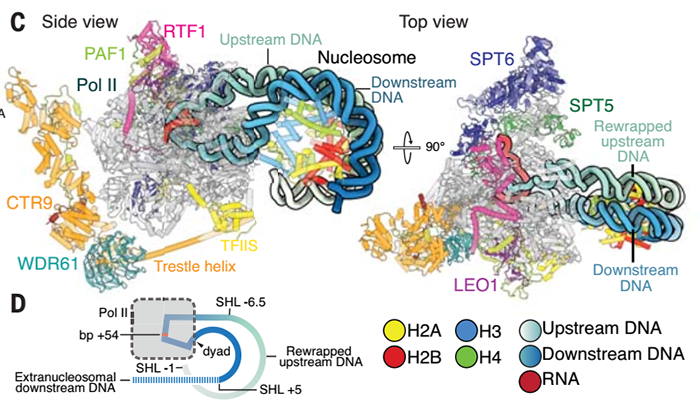
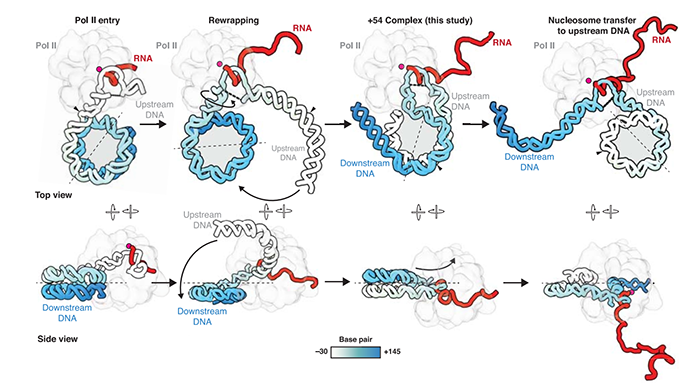
为探究Pol II如何将DNA从核小体上解开从而允许转录,及DNA随后如何再次缠绕在重建的核小体上,哈佛医学院的Lucas Farnung课题组解析出哺乳动物Pol II-延伸因子-核小体复合物的冷冻电镜结构,发现上游DNA从Pol II裂隙中漏出并在近端再次缠绕成核小体结构,揭示转录延伸过程中的核小体保留机制。结构显示Pol II和转录延伸因子在核小体保留中起直接作用,同时解释核小体保留如何阻止转录活跃基因中染色质结构的打破。上述该结果以Structural basis of nucleosome retention during transcription elongation为题发表在Science杂志。作者设计出一个核小体底物,允许Pol II转录靠近核小体二分位点(dyad),其在之前研究中被认为是Pol II的主要停留位点【11】。一段RNA引物与悬在入口处的3’-DNA退火结合,形成包含9个核苷酸的DNA•RNA杂交链从而允许Pol II继续在引物上添加核苷酸。单个核小体组装在DNA链上,随后依次添加:(1)纯化的野猪(Sus scrofa)来源的Pol II和人源延伸因子PAF1c;(2)DRB-敏感性诱导因子(DSIF),含SPT4和SPT5亚基;(3)SPT6、TFIIS、激酶-正性转录延伸因子b(P-TEFb)及3’脱氧腺嘌呤5’-三磷酸。添加UG两种核糖核苷酸可起始转录反应,3’脱氧腺嘌呤5’-三磷酸会使复合体稳定停留在改造过的停留位点,同时也作为P-TEFb的底物促进磷酸化依赖的复合物形成。转录反应产物经过分子筛纯化,获得Pol II-DSIF-PAF1c-SPT6-TFIIS-核小体复合物。洗脱产物经戊二醛适度交联后用于冷冻电镜分析。解析出的复合物结构整体分辨率为3.0Å(图1),其中核小体、Pol II和PAF1复合体的局部分辨率介于2.9-11.1 Å,进一步将已发表结构匹配到密度图中进行局部调整。重建的结构中DNA密度由上游延伸至核小体。相较于此前获得的延伸复合物,Pol II裂隙中的下游DNA位置发生变化,于是作者从头搭建DNA以匹配观察到的路径变化。图1 哺乳动物Pol II-DSIF-PAF1c-TFIIS-核小体复合物整体结构DNA•RNA杂交链的高分辨率及转录泡的完整结构使得核酸位置得以定位。Pol II中的DNA可以定位到核小体超螺旋定位(SHL)的-0.5位,而Pol II活性位点位于核小体+54位。Pol II处于混合状态,兼具转位前状态(核苷酸结合位点被新生RNA的3’端占据)和转位后状态(核苷酸结合位点未被占据)。+54位点之所以被Pol II选中是因为改造的停留位点位于核小体的+64位,这意味着停留在+54位是Pol II的主要障碍。转录的上游DNA以一种新路径离开Pol II裂隙并趋向核小体,而约55bp的转录后DNA再次缠绕核小体的近侧,再缠绕遵循核小体中的经典DNA构象。上游DNA稳定了近端的H2A-H2B二聚体,它们在核小体的转录保留中发挥关键作用。DNA再缠绕也影响下游DNA路径。下游DNA双链及DNA•RNA在Pol II活性位点处产生一个105°夹角,此时下游DNA位于夹子结构域和RPB2之间的裂隙中。更下游处,DNA与Pol II RPB5的下颌结构域接触。下游DNA双螺旋自下颌结构域弯折并朝向裂隙约30°,从而不再与RPB5接触。自Pol II裂隙中离开后,下游DNA发生扭曲并被组蛋白弯折。该DNA路径区别于已报道的下游DNA构象,此前报道中DNA往往呈直的B型构象【7-10】。下游DNA与再缠绕DNA间的小沟被组蛋白H3的N端39-43位残基占据。此外,作者还观察到下游DNA自SHL +5至+7位开始不再结合核小体并远离整个复合物。在Pol II上游,再缠绕的DNA替换此前由SPT4及SPT5 NGN和DSIP的KOW1结构域占据的Pol II结合位点。但SPT5的KOW2-3、KOWx-4及KOW5结构域的密度依然可见,说明SPT4和SPT5被部分占据是核小体DNA再缠绕及核小体保留必需的。与此相一致,功能基因组数据显示当Pol II通过核小体时Spt4/5的交联模式发生改变【12】。作者在结构数据中还观察到另一个Pol II-DSIF-PAF1c-SPT6-TFIIS-核小体复合体,停留在核小体的+38位,该复合体中未发生再缠绕,SPT4及SPT5的NGN和KOW1结构域可见并位于Pol II夹子处。再缠绕的核小体内存在Pol II、PAF1c CTR9亚基、核小体间的互作,稳定其构象:首先是RPB2上的K427与上游DNA的互作。K427在真核生物中高度保守,但在大肠杆菌中缺失,因此,突出结构域可能通过介导DNA再缠绕参与支持染色质转录。其次是RPB1的残基K203与下游核小体DNA的互作,这在酿酒酵母的Pol II-核小体结构中也曾观察到。第三,CTR9支架螺旋C末端位于靠近再缠绕DNA的SHL -1位,稳定再缠绕DNA。上述Pol II与转录延伸因子间的互作说明转录延伸机器通过多组互作稳定核小体DNA并支持核小体保留。以上数据支持Pol II通过促进DNA再缠绕到组蛋白表面帮助核小体在转录过程中保留。这种再缠绕和模板环化支持0-loop假说。该模型指出Pol II转录核小体时,其上下游的DNA仍结合在核小体上。本研究解析的结构显示loop尺寸约90bp,这与原子力显微镜测量结果近似。通过定位DNA并与经典结构比较,此前呈线性的进入侧DNA再缠绕在核小体会导致上游核小体在转录后移位,这种逆向运动在此前研究中也曾观察到。此前的结构研究显示【9,10】,稳定近端的H2A-H2B二聚体可以通过反式结合另一分子外来DNA实现。但在本研究中结构,上游DNA的再缠绕也可通过顺式的方式缠绕,即同一分子DNA再缠绕在其组蛋白核心上,说明转录过程中核小体并未完全解聚。作者由此提出一个模型(图2):当Pol II转录通过dyad时,可能导致约50bp的下游DNA完全解开,但仍缠绕在核小体的SHL -0.5至+5位。下游DNA的释放会导致核小体完全向上游DNA移位。此时,Pol II仅转录线性的下游DNA,这可以解释远端核小体为何不产生任何转录障碍。在位移过程中,组蛋白八聚体的近端和远端发生交换。而上游DNA的再缠绕可能缩短转录的核小体与前一个核小体间的距离。核小体对转录保真性发挥关键作用,一方面限定转录起始于启动子区,另一方面借助表观修饰维持转录记忆。本研究解析的哺乳动物Pol II-延伸因子-核小体复合体结构为解释转录延伸过程中核小体如何保留提出一种可能机制,同时也为染色质环境下的转录机器工作方式提供新的启示。https://www.science.org/doi/10.1126/science.abo3851